La compleja red de mecanismos de regulación de la expresión génica
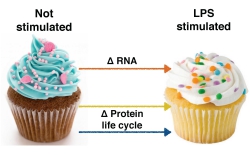
Los lipopolisacáridos (LPS) presentes en la superficie externa de microorganismos patógenos desencadenan respuestas inmunitarias potentes y rápidas mediante su interacción con células inmunitarias, especialmente con células presentadoras de antígenos como las células dendríticas (CD). Los investigadores de EXPRESSION DYNAMICS estudiaron el mecanismo de control de la respuesta inmunitaria en CD de ratón estimuladas con LPS de microorganismos patógenos. La evaluación de los niveles de expresión de ARN y de la dinámica del metabolismo de las proteínas (síntesis y degradación) proporcionó una mejor compresión general de los cambios en la expresión génica durante esta respuesta inmunitaria. Marko Jovanovic, investigador principal del proyecto, comenta: «Nuestro objetivo era crear un modelo holístico que nos permitiera determinar la contribución específica de cada uno de los distintos niveles de regulación relacionados con la expresión de cada gen». Un nuevo modelo: la guinda del pastel El resultado del trabajo del equipo de EXPRESSION DYNAMICS es un modelo en el que nuevas funciones celulares, necesarias por ejemplo para la respuesta inmunitaria aguda, son regidas principalmente a través del control a nivel de la expresión del ARN. Sin embargo, el proteoma preexistente también se modifica para adaptarse al nuevo estado celular activado, un proceso alcanzado mediante la regulación del metabolismo proteico, esto es, de la síntesis y/o de la degradación de proteínas. «Llamamos a este modelo el modelo cupcake, en el que la parte más llamativa y que frecuentemente recibe más atención es el glaseado y, en este contexto, los cambios en el glaseado son análogos a la parte ajustada por la regulación del ARNm. La parte de pastel, también importante aunque despreciada injustamente, es análoga a los genes constitutivos, que también necesitan cambiar, y, según nuestros resultados, implica el metabolismo de las proteínas», explica Marko Jovanovic. Actualmente, los investigadores están empleando el modelo de biología de sistemas para evaluar la estimulación de células nerviosas y determinar los mecanismos responsables de la regulación espacio-temporal de diferentes clases de genes en el soma y las dendritas de neuronas estimuladas. Las técnicas proteómicas y transcriptómicas utilizados para estudiar CD estimuladas por LPS permitirán a los investigadores identificar miles de genes de manera simultánea siempre y cuando los cambios proteicos estén regulados de manera diferencial en compartimentos celulares específicos como, por ejemplo, el soma y las dendritas de las neuronas. Identificar los genes responsables de la regulación Tradicionalmente, el cribado de genes inactivos se ha empleado para descubrir los principales reguladores de un proceso biológico de interés. Aunque este método ya se usa desde hace décadas en organismos modelo simples como la levadura, su aplicación en animales mamíferos modelo es mucho más difícil, máxime en líneas celulares, y frecuentemente implica fuertes lecturas fenotípicas como la supervivencia. En cambio, el equipo de EXPRESSION DYNAMICS desarrolló una nueva técnica de cribado del genoma completo basado en marcadores para identificar los reguladores de los cambios en la expresión de genes de interés. Se trata de un método más adecuado que permite llevar a cabo un cribado genético en casi todos los procesos biológicos de interés. Empleado el cribado del genoma completo modificado basado en CRISPR en CD estimuladas por LPS, los investigadores descubrieron numerosos reguladores de la respuesta inmunitaria inducida por LPS previamente desconocidos. Seguidamente, clasificaron los genes en módulos funcionales con distintos efectos en la respuesta inmunitaria frente a LPS. Aplicaciones clínicas futuras de la investigación del mecanismo de regulación génica La presentación de una solicitud de patente para la administración, el uso y las aplicaciones terapéuticas de los sistemas y la modelización CRISPR-CAS resalta el enorme alcance de esta tecnología. La identificación de genes implicados en respuestas inmunitarias mediadas por leucocitos y la modelización de alteraciones en el modo de acción de estas células así como las enfermedades asociadas son quizás los usos más obvios. El siguiente paso consistirá en evaluar sus posibles aplicaciones en quimioterapia y/o terapia génica, con el potencial de constituir una herramienta de gran utilidad para el diseño de nuevas estrategias de tratamiento. Marko Jovanovic resume su potencial repercusión en la investigación biomédica: «Siempre que exista un marcador detectable, este cribado del genoma completo basado en marcadores en células primarias de mamíferos nos permitirá emplear pruebas genéticas no sesgadas en casi todos los sistemas disponibles y, posiblemente, identificar dianas de interés para el tratamiento de enfermedades».